Qu'est-ce que GLM dans R?
Les modèles linéaires généralisés sont un sous-ensemble de modèles de régression linéaire et prennent en charge efficacement les distributions non normales. Pour cela, il est recommandé d'utiliser la fonction glm (). GLM fonctionne bien avec une variable lorsque la variance n'est pas constante et distribuée normalement. Une fonction de liaison est définie pour transformer la variable de réponse pour l'adapter au modèle approprié. Un modèle LM est fait à la fois avec la famille et la formule. Le modèle GLM a trois composants clés appelés aléatoire (probabilité), systématique (prédicteur linéaire), composant de lien (pour la fonction logit). L'avantage d'utiliser glm est qu'ils ont la flexibilité du modèle, pas besoin de variance constante et ce modèle correspond à l'estimation du maximum de vraisemblance et à ses ratios. Dans cette rubrique, nous allons en apprendre davantage sur GLM dans R.
Fonction GLM
Syntaxe: glm (formule, famille, données, poids, sous-ensemble, Start = null, model = TRUE, method = ””…)
Ici, les types de familles (incluent les types de modèles) incluent binomial, Poisson, Gaussian, gamma, quasi. Chaque distribution effectue un usage différent et peut être utilisée dans la classification et la prédiction. Et lorsque le modèle est gaussien, la réponse doit être un véritable entier.
Et lorsque le modèle est binomial, la réponse doit être des classes avec des valeurs binaires.
Et lorsque le modèle est Poisson, la réponse doit être non négative avec une valeur numérique.
Et lorsque le modèle est gamma, la réponse doit être une valeur numérique positive.
glm.fit () - Pour adapter un modèle
Lrfit () - indique un ajustement de régression logistique.
update () - aide à mettre à jour un modèle.
anova () - c'est un test facultatif.
Comment créer GLM dans R?
Nous verrons ici comment créer un modèle linéaire généralisé facile avec des données binaires en utilisant la fonction glm (). Et en continuant avec l'ensemble de données Trees.
Exemples
// Importation d'une bibliothèquelibrary(dplyr)
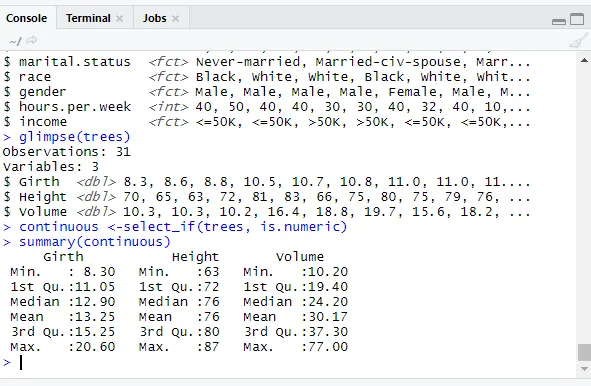
glimpse(trees)

Pour voir les valeurs catégorielles, des facteurs sont attribués.
levels(factor(trees$Girth))
// Vérification des variables continues
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Inclusion d'un jeu de données d'arbre dans la recherche R Pathattach (arbres)
x<-glm(Volume~Height+Girth)
x
Production:
| Appel: glm (formule = Volume ~ Hauteur + Circonférence)
Coefficients: (Interception) Hauteur circonférence -57, 9877 0, 3339 4, 7082 Degrés de liberté: 30 au total (c.-à-d. Nul); 28 résiduel Null Deviance: 8106 Déviance résiduelle: 421, 9 AIC: 176, 9 |
summary(x)
| Appel:
glm (formule = Volume ~ Hauteur + Circonférence) Résidus de déviance: Min 1Q Médian 3Q Max -6, 4065 -2, 6493 -0, 2876 2, 2003 8, 4847 Coefficients: Estimer Std. Erreur t valeur Pr (> | t |) (Interception) -57, 9877 8, 6382 -6, 713 2, 75e-07 *** Hauteur 0, 33393 0, 1302 2, 607 0, 0145 * Circonférence 4.7082 0.2643 17.816 <2e-16 *** - Signif. codes: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Le paramètre de dispersion pour la famille gaussienne est de 15.06862) Déviance nulle: 8106.08 sur 30 degrés de liberté Déviance résiduelle: 421, 92 sur 28 degrés de liberté AIC: 176, 91 Nombre d'itérations de Fisher: 2 |
La sortie de la fonction récapitulative donne les appels, les coefficients et les résidus. La réponse ci-dessus montre que le coefficient de hauteur et de circonférence ne sont pas significatifs car leur probabilité est inférieure à 0, 5. Et il existe deux variantes de déviance nommées nulles et résiduelles. Enfin, le score de Fisher est un algorithme qui résout les problèmes de maximum de vraisemblance. Avec le binôme, la réponse est un vecteur ou une matrice. cbind () est utilisé pour lier les vecteurs de colonne dans une matrice. Et pour obtenir les informations détaillées du résumé d'ajustement est utilisé.
Pour faire Comme test de capot, le code suivant est exécuté.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
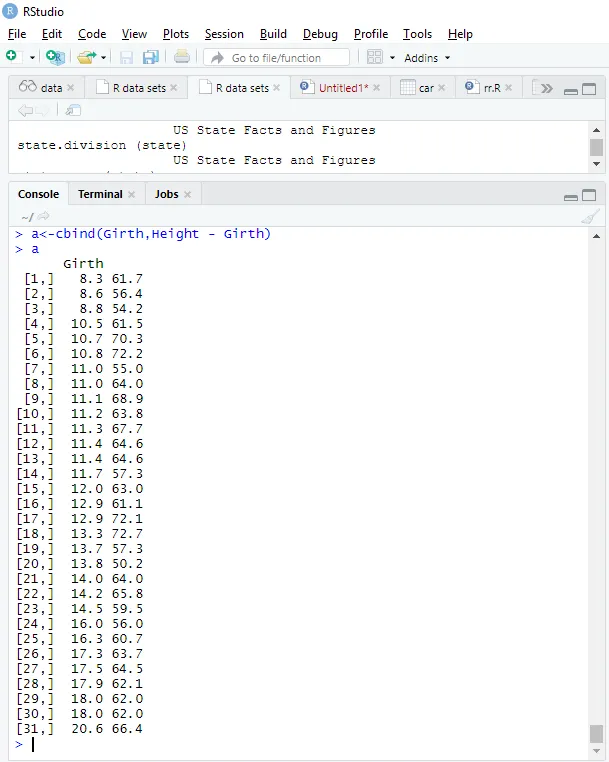
Ajustement du modèle
a<-cbind(Height, Girth - Height)
> a
résumé (arbres)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Pour obtenir l'écart type approprié
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Ensuite, nous nous référons à la variable de réponse de comptage pour modéliser un bon ajustement de réponse. Pour calculer cela, nous utiliserons l'ensemble de données USAccDeath.
Entrons les extraits suivants dans la console R et voyons comment le nombre d'années et le carré de l'année sont exécutés sur eux.
data("USAccDeaths")
force(USAccDeaths)
// Analyser l'année 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Appel:
glm (formule = nombre ~ année + annéeSqr, famille = "poisson", données = disque) Résidus de déviance: Min 1Q Médian 3Q Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Coefficients: Estimer Std. Erreur valeur z Pr (> | z |) (Interception) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** année -7.207e-03 2.354e-04 -30.62 <2e-16 *** yearSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. codes: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Paramètre de dispersion pour la famille de Poisson pris à 1) Déviance nulle: 7357, 4 sur 71 degrés de liberté Déviance résiduelle: 6358, 0 sur 69 degrés de liberté AIC: 7149.8 Nombre d'itérations de Fisher: 4 |
Pour vérifier le meilleur ajustement du modèle, la commande suivante peut être utilisée pour trouver
les résidus pour le test. D'après le résultat ci-dessous, la valeur est 0.
1 - pchisq(deviance(a1), df.residual(a1))
Utilisation de la famille QuasiPoisson pour la plus grande variance dans les données données
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Appel:
glm (formule = count ~ year + yearSqr, family = "quasipoisson", données = disque) Résidus de déviance: Min 1Q Médian 3Q Max -22, 4344 -6, 4401 -0, 0981 6, 0508 21, 4578 Coefficients: Estimer Std. Erreur t valeur Pr (> | t |) (Interception) 9.187e + 00 3.417e-02 268.822 <2e-16 *** année -7.207e-03 2.261e-03 -3.188 0.00216 ** yearSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (Le paramètre de dispersion pour la famille quasipoisson est supposé être 92.28857) Déviance nulle: 7357, 4 sur 71 degrés de liberté Déviance résiduelle: 6358, 0 sur 69 degrés de liberté AIC: NA Nombre d'itérations de Fisher: 4 |
La comparaison de Poisson avec la valeur AIC binomiale diffère considérablement. Ils peuvent être analysés par précision et rapport de rappel. L'étape suivante consiste à vérifier que la variance des résidus est proportionnelle à la moyenne. Ensuite, nous pouvons tracer en utilisant la bibliothèque ROCR pour améliorer le modèle.
Conclusion
Par conséquent, nous nous sommes concentrés sur un modèle spécial appelé modèle linéaire généralisé qui aide à focaliser et à estimer les paramètres du modèle. Il s'agit principalement du potentiel d'une variable de réponse continue. Et nous avons vu comment glm s'adapte aux packages intégrés R. Ce sont les approches les plus populaires pour mesurer les données de comptage et un outil robuste pour les techniques de classification utilisées par un scientifique des données. Le langage R, bien sûr, aide à faire des fonctions mathématiques compliquées
Articles recommandés
Ceci est un guide de GLM dans R. Ici, nous discutons de la fonction GLM et comment créer GLM dans R avec des exemples d'ensembles de données d'arborescence et une sortie. Vous pouvez également consulter l'article suivant pour en savoir plus -
- Langage de programmation R
- Architecture Big Data
- Régression logistique en R
- Emplois Big Data Analytics
- Régression de Poisson en R | Implémentation de la régression de Poisson